Por Gigi y Andrés.

La gráfica anterior es una adaptación en R de está otra gráfica que presentó hace algunos días el periódico más importante de China, Diario del Pueblo, 人民日报, 🏩 (ver acá)
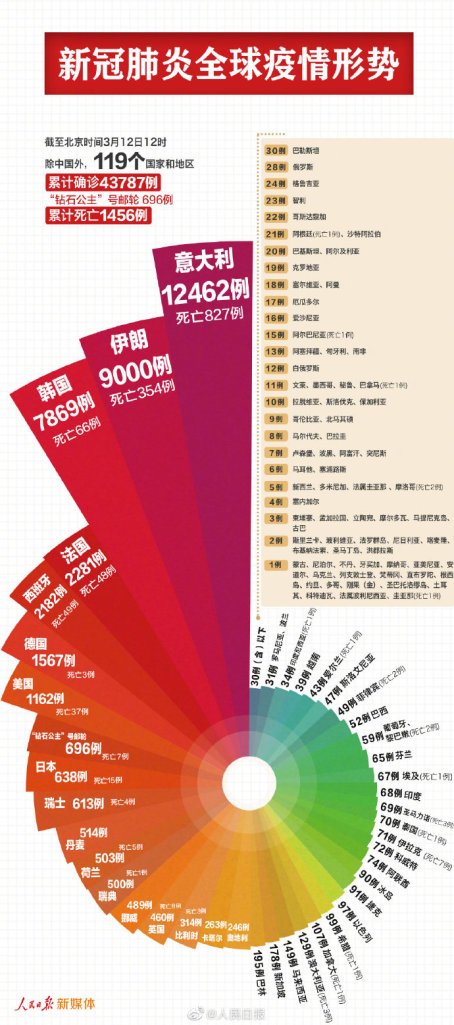
Desde hace un par de años, cada vez que veo alguna gráfica de datos, siempre me pregunto: ¿cómo se hizo? y ¿dónde están los datos para replicarla? Siendo no muy experta en gráficas sofisticadas, naturalmente busqué la solución en internet. Y la encontré: alguien de alias Red Queen Scholar publicó en su blog de weixin (o sea WeChat, o sea el guazá chino) una réplica bastante acertada. Este personaje cobra por la lectura completa, afortunadamente no mucho, así que pagué 6 yuanes (0.85 dólares) y tuve acceso a los códigos completos de R. Acabo de darme cuenta que el autor ya eliminó el cobro en esa página, así que todos pueden acceder gratuitamente en esta página. Adaptando estos códigos, fue posible obtener la gráfica en el ambiente de tidyverse en R, específicamente con ggplot2.
Lo que voy a hacer aquí es explicar un poco los códigos para los que tienen dificultad con el idioma chino (estimo que el 99.99% de los lectores de este blog lo hacen, el restante 0.01% soy yo 😝).
- Empecemos con los datos a utilizar, Red Queen Scholar utilizó los datos de la librería nCov2019 que a su vez se alimenta del portal de noticias chino Tencent. Pero prefiero usar los datos de la Universidad Johns Hopkins, los del famoso dashboard que seguramente ustedes han visto ya. Así que empiezo descargando los datos disponibles en GitHub. Los datos de casos confirmados y de fallecidos están en formato csv en dos direcciones diferentes.
- En los dos archivos están los datos por país/región desde el 22 de enero hasta el presente, pero solo necesito los del último día, así que eliminamos datos pasados y solo dejamos las primeras dos columnas sobre ubicación y la última columna.
- Ahora se procede a calcular el número de casos y de fallecidos por país, los datos vienen por regiones, pues países como China o los Estados Unidos son grandes (geográficamente hablando) y los datos están desagregados a nivel de provincias o estados.
- Luego se conforma una sola base de datos con el número de casos y el de fallecidos. También se cambia el nombre de algunos países para que quepan en la gráfica.
- Ustedes pueden ver que para las barras más chicas (con letras negras) se muestra el número de casos y, en paréntesis, el número de fallecidos. Adicionalmente, hay países que tienen el mismo número de casos, por ejemplo, India y San Marino ambos tienen 62 casos, y son representados por una misma barra. Entonces necesitamos crear todos estos rótulos para agregarlos en la gráfica.
- Es claro que en la gráfica no caben todos los países, así que escogeremos solo algunos. ¿Cuáles?, pues los que más casos de coronavirus tienen. Se utilizarán los 40 países con más casos para la gráfica, y finalmente se añade una variable angle para los ángulos de las barras. También se arregla el nombre de los países, y finalmente tenemos la base de datos para la gráfica.
- Como ustedes seguramente saben, las gráfica de ggplot son por capas: creamos una hoja blanca con ggplot y le vamos poniendo capas de barras, líneas, textos, números, títulos, etc. Todas estas capas se unen con el signo +, y forman una sola instrucción, así que las explicaciones las encuentran dentro del código.
Acá va el código final. Pueden copiar y pegar para obtener la gráfica de forma automática.
rm(list=ls())
library(tidyverse)
library (readr)
urlfile.death="https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Deaths.csv"
urlfile.conf="https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_19-covid-Confirmed.csv"
death <- read_csv(url(urlfile.death))
conf <- read_csv(url(urlfile.conf))
colnames(death) <- gsub(" |/", '.', colnames(death))
colnames(conf) <- gsub(" |/", '.', colnames(conf))
# Se deja solo la fecha del último día
fecha <- "3.11.20"
p <- which(colnames(conf)==fecha)
death <- death[, c(1,2,p)]
conf <- conf[, c(1,2,p)]
# Calcular el número total de casos confirmados por país
names(death)[3]
names(death)[3] <- "today"
names(death)[2] <- "country"
death1 <- death %>% select(-Province.State) %>%
group_by(country) %>%
summarise(death = sum(today))
# Calcular el número total de fallecidos por país
names(conf)[3] <- "today"
names(conf)[2] <- "country"
conf1 <- conf %>% select(-Province.State) %>%
group_by(country) %>%
summarise(conf = sum(today))
# Unir casos confirmados y fallecidos
x <- full_join(death1, conf1, by = "country")
# cambiar algunos nombres largos por unos más cortos
x$country <- recode(x$country, 'Korea, South' = 'S.Korea', 'United Kingdom' = 'U.K.')
# Crear una variable country_dead que tenga el nombre del país y el número de fallecidos
x$country_dead <- c(ifelse(x$death == 0,print(x$country),paste(x$country," (",x$death," D)",sep = "")))
# juntar los países según el número de casos
x1 <- x %>% group_by(conf) %>% summarise(country = paste(country,collapse = ", "))
# juntar los países según el número de casos, e incluye el número de muertes
x2 <- x %>% group_by(conf) %>% summarise(country_dead = paste(country_dead,collapse = ", "))
# número de muertes para los países agrupados
x3 <- x %>% group_by(conf) %>% summarise(death = paste(death,collapse = ", "))
x4 <- merge(x1,x2)
x5 <- merge(x4,x3)
x5 <- as.data.frame(x5)
# x5 es la base final
x5 <- x5 %>% arrange(desc(conf))
# Elegir los 40 paises con más casos confirmados
k <- 40 # número de paises a graficar
top40 <- x5[1:k,]
# algunas cosas necesarias para la gráfica
top40$country <- factor(top40$country,levels = top40$country)
top40$angle <- 1:k*360/k
# Ahora sí, la gráfica
ggplot(top40,aes(x = country, y = conf, fill = country)) +
# Gráfica de barras
geom_bar(stat = "identity",position = "stack",width = 1) +
# Limipar informaciones que sobran
theme_void() +
# Limpiar un poco más
theme(legend.position = "none") +
# Tomar logaritmo por la gran diferencia entre los valores, de lo contrario, muchísimos paises no se verán en la gráfica
scale_y_log10() +
# Usar coordenada polar, direction = -1 indica que la dirección es en contra de manecillas de reloj
coord_polar(direction = -1) +
# Agregar la zona circular blanco del centro
geom_bar(aes(y = I(2)),stat = "identity",width = 1,fill = "white")+
# Agregar dos aros blancos transparentes en la mitad de la gráfica
geom_bar(aes(y = I(4)),stat="identity",width = 1,fill = "white",alpha = 0.2) +
geom_bar(aes(y = I(6)),stat="identity",width = 1,fill = "white",alpha = 0.1) +
# Empieza la parte aburrida de agregar textos y números
# Colocar el número total de casos de los primeros 5 paises
geom_text(aes(label = paste(country,paste(conf,"C",sep = ""),sep = "\n"),
y = conf*.8, angle = angle),
data = top40[1:5,],color = "white",
fontface = "bold", vjust = 1, family = "Helvetica",size = c(2.6, 2.3, 2.2, 2.2,2.2))+
# Colocar el número de fallecidos de los primeros 5 paises
geom_text(aes(label = paste(death,"M",sep = ""),
y = conf*.4, angle = angle),
data = top40[1:5,],color = "white",
fontface = "bold", vjust = 4, family = "Helvetica",size = 1.5) +
# Colocar el nombre de los paises de posición 6 a la 10
geom_text(aes(label = country, y = conf*.8),
data = top40[6:10,],color = "white",
vjust = seq(1.3,0.9,length.out = 5),
hjust = c(0.4, 0.2, 0.1, 0.15, 0.1),
fontface = "bold", family = "Helvetica",
size = seq(2.3,2.1,length.out = 5)) +
# Colocar el nombre de los paises de posición 11 a la 20
geom_text(aes(label = country, y = conf*.8),
data = top40[11:20,],color = "white",
vjust = seq(0.6,0.2,length.out = 10),
hjust = c(0.2, 0.2, 0.1, 0.3, 0.1, 0.3, 0.3, 0.3, 0.4,0.4),
fontface = "bold", family = "Helvetica",
size = seq(2.2,1.3,length.out = 10)) +
# Colocar el número total de casos de los paises de posición de 6-20
geom_text(aes(label = paste(conf,"C",sep = ""),
y = conf*c(seq(.55,.45,length.out = 6),
seq(.47,.65,length.out = 9))),
data = top40[6:20,],color = "white",
vjust = c(2, 2, 2, 1.7, 1.8, seq(0.2,0,length.out = 10)),
hjust = seq(-0.2,0.5,length.out = 15),
fontface = "bold", family = "Helvetica",
size = seq(1.9,1.5,length.out = 15)) +
# Colocar el número de muertes de los paises de posición de 6-20
geom_text(aes(label = paste(death,"D",sep = ""),
y = conf*c(seq(.35,.21,length.out = 6),
seq(.22,.45,length.out = 9))),
data = top40[6:20,],color = "white",
vjust = c(2, 2.5, 2.5, 2.5,2.5, seq(0.4,0,length.out = 10)),
hjust = seq(0.2,0.5,length.out = 15),
fontface = "bold", family = "Helvetica",
size = 1.4) +
geom_text(aes(label = paste(paste(conf,"C",sep = ""),
country_dead,sep = " "),
y = conf*1.1,angle = angle+90),
data = top40[21:40,],color = "black",
vjust = 0, hjust = 0,
fontface = "bold", family = "Helvetica",size = 1.5) +
# Colocar títulos, subtítulos, etc.
labs(title = 'Coronavirus al día de hoy', subtitle='(C = casos confirmados, M = muertes)',
caption ='Datos tomados de: https://github.com/CSSEGISandData/COVID-19') +
# Apariencia de títulos, subtítulos, etc.
theme(plot.title=element_text(size=16, hjust=0.5, face="bold", vjust=-10),
plot.subtitle = element_text(size=8, hjust=0.5, face="bold", vjust=-23),
plot.caption = element_text(size=5, hjust=0.5, face="bold", color='grey', vjust=80))
setwd("/Users/psirusteam/Desktop")
ggsave("Flor.pdf")
ggsave("Flor.jpg")
Sí, muchas líneas de códigos, pero lo importante es que funcionan. Tengan en cuenta que cuando ustedes ejecuten estas líneas, no producirán la misma gráfica que mostré anteriormente, la razón es que los datos son tomados de la página de GitHub que se actualizan constantemente, entonces los datos que ustedes utilizarán no serán los mismos de los que usé para la gráfica, (correspondientes al 12 de marzo). Y precisamente por esta misma razón, es muy probable que ustedes tengan que ajustar la posición de textos o números de algunas barras, porque estos fueron agregados manualmente, o sea, mirando barra por barra en la gráfica y modificando.